Секвенирование (Генная инженерия)
Страница 3
• Смесь для ddT-терминации: по 80 мкМ dGTP, dATP, dCTP, dTTP, 8 мкМ ddTTP, 50 мМ NaCl
• Стоп-раствор: 90% формамид, 20 мМ ЭДТА, 0,05% бромфеноловый синий, 0,05% ксилолцианол
• Универсальный праймер для секвенирования - 40 (0,5 пмоль/ мкл)
• [35S]dATP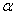 S (1 мКи/37МБк в 100 мкл) (Amersham, UK; в состав набора не входит)
S (1 мКи/37МБк в 100 мкл) (Amersham, UK; в состав набора не входит)
• 0,1 М ДТТ
Методика
Все реактивы добавляют с помощью диспенсера на 2 мкл Hamilton (PB600), соединенного с адаптером и шприцом 1710 с газовым затвором. Смесь для мечения предварительно разбавляют в пять раз.
1. Для каждой секвенируемой матрицы смешивают в микроцен-трифужной пробирке на 1,5 мл для получения праймерной смеси 6 мкл воды, 1 мкл универсального праймера и 2 мкл реакционного буфера.
2. Размечают микроплашку Falcon 3911. В верхней ее части наносят номера клонов, а слева, сверху вниз, — буквы TCGA.
3. На дно каждой ячейки наносят 2 мкл праймерной смеси, на боковые стенки — по 2 мкл раствора секвенируемой матрицы и центрифугируют плашку. Накрывают ее пленкой Saran® и крышкой и помещают в водяную баню с температурой 70°С на 5 мин. Охлаждают плашку на столе (за это время происходит отжиг праймера и ДНК М13).
4. Пока плашка охлаждается, готовят смесь для мечения. Для этого в микроцентрифужную пробирку на 1,5 мл вносят 0,5 мкл 35S-dATP, 1 мкл 0,1 М ДТТ, 2 мкл разведенной смеси для мечения и 3,5 мкл воды.
5. Размечают поликарбонатную микроплашку Techne 96® так же, как первую плашку, и в ячейки в ряду "Т" вносят по 2 мкл смеси для ddT-терминации. Аналогичным образом вносят смесь для терминации в ячейки остальных рядов и помещают плашку в термостат для микроплашек с температурой 42°С.
6. После охлаждения плашки (п. 3) в течение 30 мин добавляют к смеси для мечения (для каждой матрицы) последовательно 1,77 мкл буфера для разведения фермента и 0,22 мкл фермента Sequenase® II. (Это позволяет держать фермент Sequenase® II вне холодильника минимальное время.)
7. По 2 мкл этой смеси наносят на боковую стенку ячеек, содержащих праймерную смесь, и центрифугируют плашку для перемешивания компонентов. Включают секундомер.
8. Через 2 мин начинают переносить раствор из ячеек первой плашки в соответствующие ячейки предварительно нагретой и помещенной в термостат поликарбонатной плашки. Для этого используют обычную микропипетку, быстро меняя наконечники после каждой ячейки (помните, что использованные наконечники радиоактивны).
9. После того как перенесен раствор из последней ячейки, включают секундомер и в наконечник на шприце Hamilton набирают стоп-раствор.
10. Через 5 мин наносят по 5 мкл стоп-раствора на боковую стенку каждой ячейки и центрифугируют плашку. После центрифугирования плашку, закрытую крышкой, можно хранить в морозильнике до использования (при - 20°С 35S-продукты можно хранить в течение недели).
Амплифицированные последовательности нуклеотидов можно увидеть в УФ-свете после фракционирования продуктов ПЦР с помощью гель-электрофореза вприсутствии бромистого этидия. В большинстве случаев после ПЦР при наличии 1 - 10 нг ДНК-матрицы выявляется только одна полоса ДНК ожидаемой электрофоретической подвижности. Чувствительность и специфичность детекции продуктов амплификации значительной увеличиваются при использовании различных вариантов ДНК—ДНК-гибридизации с олигонуклеотидами-зондами, имеющими радиоактивную биотиновую, флюоресцентную или хемолюминесцентную метку. Это сделало возможным проведение работ с минимально возможным количеством материала, (например, с одной клеткой, одной копией гена) без предварительной его очистки.
В качестве исходной матрицы для ПЦР может быть использована ДНК (или кДНК, полученная с помощью предварительной обратной транскрипции РНК), выделенная как из свежеполученных клеток и тканей, так и из замороженных, высушенных или фиксированных препаратов, имеющих частично деградированные нуклеиновые кислоты, т. е. объекты, ранее недоступные для анализа. Так, с помощью методов ПЦР была амплифицирована, клонирована и секвенирована ДНК египетской мумии, продемонстрирована возможность анализа специфических участков ДНК при наличии одного волоса, клетки, сперматозоида в целях идентификации личности и пола хозяина.
Серповидно-клеточная анемия,  -талассемия, диабет, ревматоидный артрит, мышечная дистрофия, фенилкетонурия, гемофилия, дефицит
-талассемия, диабет, ревматоидный артрит, мышечная дистрофия, фенилкетонурия, гемофилия, дефицит 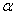 -антитрипсина - вот далеко не полный список генетических заболеваний, которые могут быть выявлены на ранних стадиях развития эмбриона с помощью ПЦР Разработаны также подходы к раннему выявлению и прогнозированию онкологических заболеваний.
-антитрипсина - вот далеко не полный список генетических заболеваний, которые могут быть выявлены на ранних стадиях развития эмбриона с помощью ПЦР Разработаны также подходы к раннему выявлению и прогнозированию онкологических заболеваний.
3. ФИЛОГЕНЕТИЧЕСКИЙ АНАЛИЗА ГЕНОМОВ ВИРУСОВ.
Филогенетический анализ молекулярных данных является одним из подходов к теоретическому изучению структуры и функции генетических макромолекул (РНК, ДНК, белков) и их эволюционного преобразования. Основная цель филогенетического анализа - изучение эволюционного порядка дивергенции последовательностей генов и белков или их частей, а также восстановление списков эволюционных событий (замен нуклеотидов, делеций и вставок) в предковых линиях этих макромолекул.
Основным инструментом филогенетического анализа является сравнение близких по структуре или по функции генов или белков, и прежде всего, сравнение их первичных последовательностей.
Важнейшим свойством функционально значимых структур макромолекул является их эволюционный консерватизм. Чем меньше функциональная важность отдельных участков генов, тем больше они имеют тенденцию к эволюционной изменчивости. Так, например, псевдогены по-видимому полностью утратили функциональную активность. Для них характерно быстрое накопление в ходе эволюции различных замен, делеций и вставок, разрушающих исходную структуру гена. С другой стороны, гистоны Н4, играющие важную роль в упаковке хроматина, почти не изменялись на протяжении всей эволюции животных.
Консервативность генов позволяет выявить отдаленное родство между их представителями, давно разошедшимися в ходе эволюции и выполняющими иногда разные функции. Однако для филогенетического анализа необходимо и наличие определенного уровня изменчивости генов. Мутации, делеции и вставки являются своего рода метками, благодаря которым удается восстановить пути эволюции современных форм макромолекул. Гены с разной величиной консервативности пригодны для изучения разных эволюционных уровней. Сильно консервативные гены и их продукты (гистоны, тРНК) нельзя , например, использовать для исследования эволюции отрядов и более мелких таксонов, но с успехом можно применять для изучения эволюции более крупных таксонов. Сильно вариабельные гены, наоборот, дают хорошее разрешение лишь на поздних эволюционных этапах.
В последнее время метод полимеразной цепной реакции (ПЦР) с последующим анализом нуклеотидной последовательности широко используется для точной идентификации вирусов и определения их родства в отношении других штаммов. Для сравнительной характеристики геномов различных штаммов вирусов также проводят рестрикционный анализ ПЦР-продуктов. Обычно для построения филогенетического дерева используются данные последовательностей нуклеиновых кислот. Филогенетическое дерево очень ясно показывает родство между вирусами, если анализируется большое количество изолятов. Для этих целей существует много компьютерных программ. Наиболее популярные пакеты программ- PHYLIP(PHYLogeny Inference Package), PAUP(Phylogentic Analysis Using Parsimong),CLUSTAL и MEGA.
Для филогенетического анализа особенно интересны РНК-содержащие вирусы, которые существуют как гетерогенные популяции. Их геном более генетически пластичен, чем геном ДНК-содержащих вирусов.
Так, геном вируса бешенства, который относится к роду Lyssavirus семейства Rhabdoviridae, представлен одноцепочечной негативной РНК длиной около 12000 пар оснований (п.о) , кодирующей пять основных белков.